上海交大研究团队面向真实语境,定量解析“非编码突变→基因表达”,助力复杂疾病研究
时间:2025-10-08
来源:上海交大 2025-10-08 17:13
这项工作试图回答临床与科研界长期的两大难题:一个非编码突变,会让目标基因“上调还是下调”?影响到底有多强? 更关键的是,答案会随组织、细胞类型甚至疾病状态而改变。在医学的语境下,越来越多的证据表明:大量致病线索潜伏在基因组 非编码区 。但临床与基础研究真正需要回答的,是两个更 落地 的问题 某个非编码突变会让下游基因上调还是下调?以及这种影响在不同组织或细胞类型中是否一致、强弱如何?近日,围绕这两个关键点,上海交通大学生物医学工程学院林关宁教授团队提出了EMO研究框架并发表于Nature Computational Science。这项工作试图回答临床与科研界长期的两大难题:一个非编码突变,会让目标基因 上调还是下调 ?影响到底有多强? 更关键的是,答案会随组织、细胞类型甚至疾病状态而改变。EMO 的设计初衷,就是把这种 语境决定效应 的规律写进模型里,让结论可迁移、可解释,也更接近真实生物学。

为什么这很难?传统深度学习方法多以DNA 序列为唯一输入,得到的是面向 平均个体 的静态结论:同一变异在肝脏与脑内可能方向相反、在炎症刺激前后强弱迥异,但旧模型很难识别。EMO在输入端把DNA 序列与 ATAC-seq 染色质可及性逐碱基对齐后联合建模;在结构端采用 双分支+分而治之 思路:一支聚焦变异点附近的局部影响,另一支覆盖从突变位点到靶基因 TSS 最远 1 Mb 的长距离顺式调控区间;在任务端把方向判别(上/下调)与强度回归(eQTL 斜率)拆分训练,并用稀疏注意力与尺度感知池化,既压住了长序列计算量,又把增强子、TF 结合位点等功能区域 高亮 出来。这套机制既像 广角镜头 ,也像 放大镜 ,兼顾远近两端的调控证据。

图1. EMO模型架构及应用场景
训练素材同样强调 语境 。团队以 GTEx v8 多组织 eQTL 为标签,配对 EpiMap 的组织/细胞类型特异 ATAC-seq 数据,序列与表观信号逐碱基对齐后输入模型,大幅增强了 不同组织/细胞里同一突变可能不同 的可学习性。这样做的结果,是在看不见的组织也能基于其 ATAC-seq 进行 零样本 推断;而在小样本目标组织,只需少量微调即可获得稳定输出,避免端到端小样本训练常见的 全部判上调 的坍塌。
从大家关心的 到底好用吗 开始看数据。首先,在多组织独立测试中,带跨组织预训练的 EMO-zeroshot/finetune 相比端到端与多款代表性方法(如 Enformer、Basenji2、Expecto)整体表现更稳,尤其在上/下调方向判别上优势明显,说明模型确实学到了 序列 染色质 表达 的通用表征而不是死记硬背。
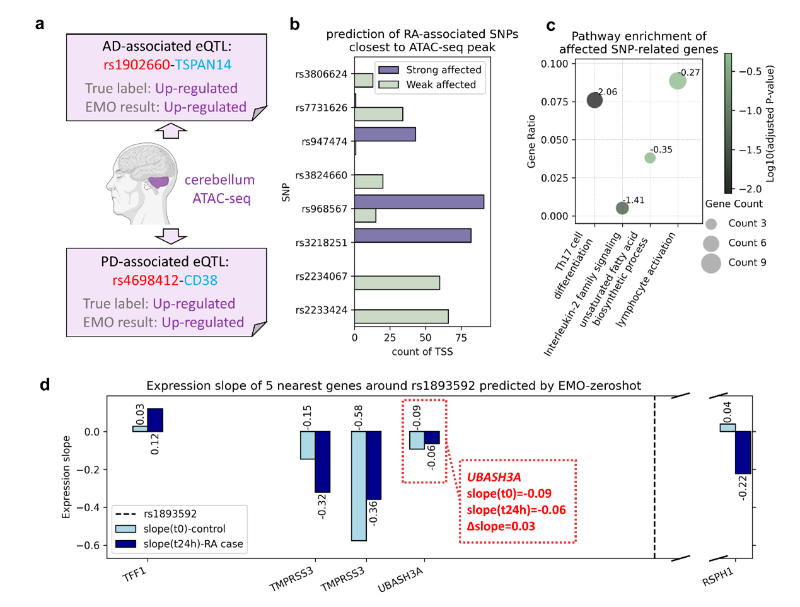
图2. EMO在疾病关联SNP分析中展示出了Zero-Shot预测能力与疾病动态预测能力
其次,把模型迁移到脑组织(MetaBrain 的海马体与脊髓)时,在样本有限的真实条件下,微调后的 EMO 在 10 100 kb 中距区间把 AUC 分别提升 0.164 与 0.079,并有效避免预测坍塌,这对难获取样本的组织尤其关键。
第三,EMO 进一步下沉到单细胞层面。在 OneK1K 队列的六类细胞中,EMO 的方向判别 AUC 达到 0.861 0.948;在与相关的 rs1465697 案例里,模型能在不同 T 细胞亚群里给出细胞类型特异的强度估计(斜率),把 到底哪类免疫细胞更敏感 的问题落到量化上。对精准分型与靶点优选,这类 指向细胞类型 的证据非常实用。
更贴近应用的,是零样本推断。只要目标组织有可用 ATAC-seq,哪怕没有在该组织训练,EMO 也能直接判别方向。以小脑为例,团队对两则神经精神疾病相关 eQTL 做了验证:rs4698412 CD38()与 rs1902660 TSPAN14(),模型分别给出 93.7% 与 69% 的上调概率,方向均与文献一致。这意味着在低样本、低门槛的情形下,仍可得到可信的机制线索。
为验证区分力的 下限 ,研究者还构造了一个 近似非因果 的负控集合(PPC 0.001 且 |slope| 小),结果显示 EMO 的回归输出能显著区分强效上/下调与弱效/无效变异;更有意思的是,在这些 非因果 样本里,模型还能 捞出 若干与疾病风险相关的 位点,提示它有望补回细粒度分析的漏检。
在免疫疾病的真实场景中,EMO 还能把疾病过程前后的调控差异 量出来 。团队用 CD4⁺T 细胞 未刺激 vs 24h 刺激 的 ATAC-seq 表示(RA)的状态变化,围绕 RA 相关 GWAS 位点,计算两状态下的斜率差值( slope),据此分组并做通路富集,结果显著聚焦在Th17 分化与IL-2 家族等核心免疫通路。这条 位点 强弱差异 通路 的链路,恰是临床研究者最需要的可行动证据。
故事的 另一半 来自 Methven。今年1月,团队在Advanced Science发表了Methven工具:它回答的是 非编码突变如何改变 DNA 甲基化 ,而且是单细胞分辨率。Methven同样整合 DNA 序列与单细胞 ATAC-seq,以 DNABert2 预训练表征 + BiGRU 为核心,在 100 kb 区间内建模 SNP-CpG 作用,既做方向判别也做强度回归。系统比较显示,它在长短距离两档均优于既有方法(如 CpGenie、Enformer),对单核细胞等外部数据也有不错的外推;在 RA 应用里,Methven 能定位到与病程相关的 CpG 与通路,提供表观层的因果线索。


