中国博后一作Nature论文:开发AI模型,高精度预测酶的特异性,已回国加入南京师范大学
时间:2025-10-09
来源:生物世界 2025-10-09 15:33
该研究提出了一种结合跨注意力机制(Cross-Attention)与 SE(3)-等变图神经网络(SE(3)-equivariant GNN)的创新人工智能模型——EZSpecificity。酶(Enzyme)是生命的分子机器,其功能的一个关键特性是底物特异性 即酶能够识别并选择性地作用于特定底物的能力。这种特异性源自酶活性位点的三维(3D)结构以及反应复杂的过渡态。实际上,许多酶并非传统认为的那样 专一 ,它们可能具有多功能性,除了作用于其最初进化时涉及的底物,还能作用于其他结构相似的底物,从而催化不同类型的反应。
此外,数以百万计的已知的酶仍缺乏可靠的底物特异性信息,这阻碍了它们的实际应用以及我们对自然界生物催化多样性的全面理解。
2025 年 10 月 8 日,伊利诺伊大学厄巴纳-香槟分校(UIUC)赵惠民教授团队(崔海洋、苏雨峰为共同第一作者)在国际顶尖学术期刊Nature上发表了题为:Enzyme specificity prediction using cross attention graph neural networks的研究论文,论文第一作者崔海洋博士现为南京师范大学教授。
该研究提出了一种结合跨注意力机制(Cross-Attention)与SE(3)-等变图神经网络(SE(3)-equivariantGNN)的创新人工智能模型 EZSpecificity,可高精度预测酶的底物特异性,以帮助研究人员找到酶与底物的最佳组合。这项研究为 AI 在酶工程、合成生物学和绿色制造中的深度应用奠定了新的方法学基础。

论文通讯作者赵惠民教授表示,如果我们想要获得某种使用酶的产品,我们希望使用最佳的酶和底物组合,EZSpecificity是一种 AI 模型,它可以分析酶的序列,然后预测哪种底物最适合与该酶结合。该模型与团队两年前开发的用于从酶的序列预测其功能的CLEAN AI模型高度互补(该模型于 2023 年 5 月 发表在了Science期刊),二者共同构成了一个从 功能发现 到 应用 的完整酶研究 AI 工具链。
酶(Enzyme)是能够催化分子反应的大分子蛋白质(核酶除外),它们有口袋状的结构域,目标分子(称为底物)可嵌入其中。酶与底物的契合程度被称为特异性(specificity)。我们通常把酶与底物相互作用类比为 锁和钥匙 :只有合适的钥匙才能打开锁。但酶的功能并非如此简单。
要找出酶与底物的最佳组合,颇具挑战性,因为口袋并非一成不变。酶在与底物相互作用时,其构象会发生变化,更像是诱导契合。而且有些酶具有多态性,能够催化不同类型的反应,这使得预测变得非常困难。因此,研究团队希望利用机器学习模型和实验数据来真正证明哪种配对效果最佳。
此前,已有研究团队提出了酶特异性模型,但这些模型在准确性以及能够预测的酶促反应类型方面都存在局限性。
赵惠民团队意识到,要提高 AI 预测特异性的能力,还需要改进并扩充机器学习模型所依据的数据库。于是,他们与伊利诺伊大学Diwakar Shukla展开合作,后者针对不同类别的酶进行了对接研究,创建了一个大型数据库,其中不仅包含酶的序列和结构信息,还涵盖了不同类别酶在不同类型的底物周围所呈现的构象信息。
研究团队表示,捕捉酶与其底物相互作用的实验通常进展缓慢且复杂,所以团队进行了大量的对接模拟,以补充和拓展现有的实验数据。研究团队聚焦于酶与其底物之间的原子级相互作用,数百万次的对接计算提供了这一缺失的拼图,从而构建出一个高度准确的酶特异性预测模型 EZSpecificity。该模型使用SE(3)-等变图神经网络(SE(3)-equivariantGNN)来表征酶 底物的三维结构关系,并引入跨注意力机制(Cross-Attention)模块来强化酶的活性口袋与底物分子之间的高维信息交互。
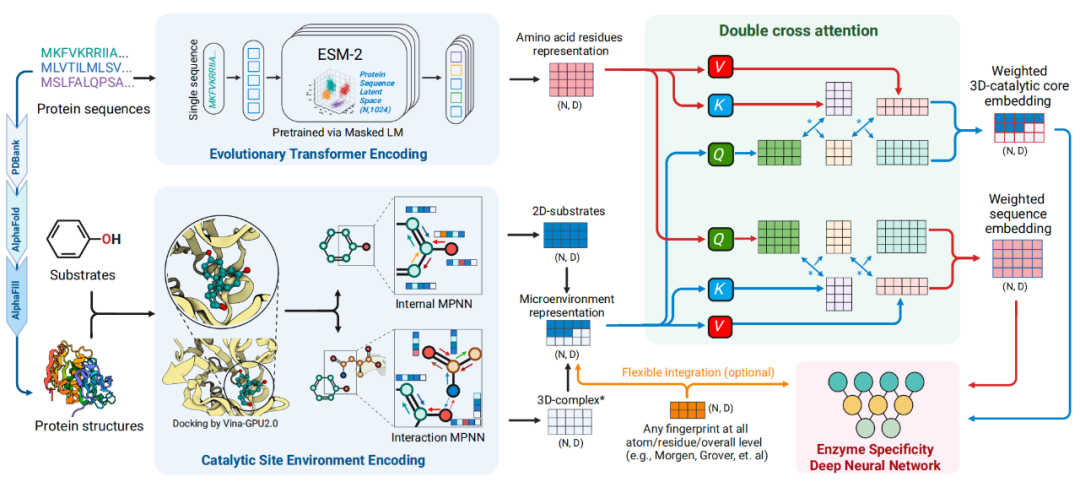
EZSpecificity模型总体框架
随后,研究团队在四个旨在模拟实际应用的场景中,将 EZSpecificity 与当前领先的模型 ESP 进行了并行测试,在所有场景中,EZSpecificity 的表现都优于 ESP。最后,研究团队通过研究 8 种卤化酶(这一类酶尚未得到充分表征,但其在合成生物活性分子方面的应用日益增多)和 78 种底物,对 EZSpecificity 进行了实验验证。结果显示,EZSpecificity 准确识别唯一反应底物的成功率高达91.7%,而 ESP 的准确率仅为 58.3%。
赵惠民教授表示,现在还不能说EZSpecificity对每种酶都能有效预测,但对于某些酶来说,该研究已经证明了其效果很好。团队希望将这个工具提供给其他研究人员使用,因此开发了一个用户界面,只需输入底物和蛋白质序列,就可以使用EZSpecificity来预测该底物是否能很好地发挥作用。
EZSpecificity模型,标志着酶研究从 智能注释 迈向 智能识别 ,为理解生物催化的分子机制、推动AI驱动的酶工程、代谢网络改造及绿色催化剂开发提供了全新的技术路径。研究团队表示,下一步计划拓展该模型,用于分析酶的选择性,即酶是否对底物上的特定位点有偏好,以帮助排除具有脱靶效应的酶。此外,还计划继续用更多的实验数据来完善 EZSpecificity。
版权声明 本网站所有注明“来源:100医药网”或“来源:bioon”的文字、图片和音视频资料,版权均属于100医药网网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:100医药网”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。 87%用户都在用100医药网APP 随时阅读、评论、分享交流 请扫描二维码下载->
